El ADN obtenido durante el ensayo de RT-PCR se puede secuenciar y por tanto podemos obtener el orden de los nucleótidos en el genoma. La secuenciación del ADN es útil para discriminar entre cepas individuales. Normalmente, la relación entre los diferentes aislados se ha realizado en base a la comparación de las secuencias de ORF5, ORF6 u ORF7.
Con respecto a las muestras de sangre individuales, es importante tener en cuenta que para secuenciar se necesitan muestras con valores de Ct por debajo de 32. Las muestras con valores de Ct> 32 son difíciles de secuenciar, ya que la cantidad de virus es insuficiente.
Los fluidos orales grupales y los pooles de sangre no deben usarse para secuenciar, ya que al mezclar diferentes animales estamos disminuyendo la cantidad de virus presente y además pueden contener fácilmente más de una cepa.
La secuenciación es una herramienta de diagnóstico complementaria. Es útil para poder tomar decisiones en relación a las medidas de control del virus.
Mediante la secuenciación es posible:
- Distinguir especies
- Distinguir aislados de virus de campo de las vacunas
- Obtener una valiosa información epidemiológica para determinar la fuente de la infección, cuántas cepas están presentes en un hato/granja/población/empresa/región, cuál es el más frecuente, los eventos de recombinación, etc.
En general, si se obtienen secuencias con cierta frecuencia (se recomienda secuenciar cada cepa involucrada en un brote o periódicamente, al menos cada seis meses), podemos llegar a determinar la fuente principal de infecciones y cómo circulan los aislados en un hato o en una región.
Eventualmente, podríamos usar esta información para establecer mejores medidas de control de la enfermedad.
Hoy en día, a través de la secuenciación no es posible:
- (En general) predecir la virulencia de un aislamiento determinado;
- Predecir el nivel de protección cruzada entre dos aislados, ya que los marcadores genéticos de virulencia y protección cruzada no se conocen con exactitud.
La secuencia no debe usarse para decidir qué vacuna debe usarse frente a un aislado concreto, ya que se ha demostrado que el nivel de protección cruzada que pueda existir entre dos cepas no está relacionado con el nivel general de homología (relación genética).
Para poder comparar el grado de similitud entre aislados y agruparlos según sus secuencias, se puede dibujar un árbol filogenético.
En general, para ilustrar las relaciones entre varios aislados los árboles son más útiles que la simple comparación de porcentajes de homología, ya que puede suceder que un grupo de cepas compartan un mismo porcentaje de similitud aun siendo diferentes.
En estos casos, los árboles son cruciales para observar las relaciones reales.
Árbol filogenético obtenido mediante el análisis de siete secuencias ORF5 de cuatro orígenes diferentes.
La barra representa una sustitución de 0,1 nucleótidos por sitio. La interpretación de este árbol es la siguiente: en la rama inferior hay un aislado no relacionado del origen 1 (O1) (O1- I3); su similitud con O1-I1 y O1-I2 del mismo origen, ubicados en la rama superior del árbol, es de 93,4% y 93,2%, respectivamente.
La similitud entre estos dos aislados relacionadas (O1-I1 y O1-I2) es del 97.5%. En el origen 2, hay dos aislados no relacionados: I4 e I5 (la similitud entre ellos es del 96,6%).
Probablemente, existe un vínculo entre los aislados O2-I4 y el origen 1 y entre O2-I5 y el origen 3. Finalmente, los aislados
I6 e I7 del origen 3 están estrechamente relacionados (> 99,8%; seguramente son la misma cepa).
Comúnmente se acepta que dos aislados son diferentes si la similitud entre ellos es inferior al 97% (ORF5).
Sin embargo, este punto de corte es completamente arbitrario. Para evitar interpretaciones erróneas, se debe tener en cuenta información adicional, tales como fechas y lugares de aislamiento, las relaciones epidemiológicas que se establecen entre las granjas y orígenes implicados.
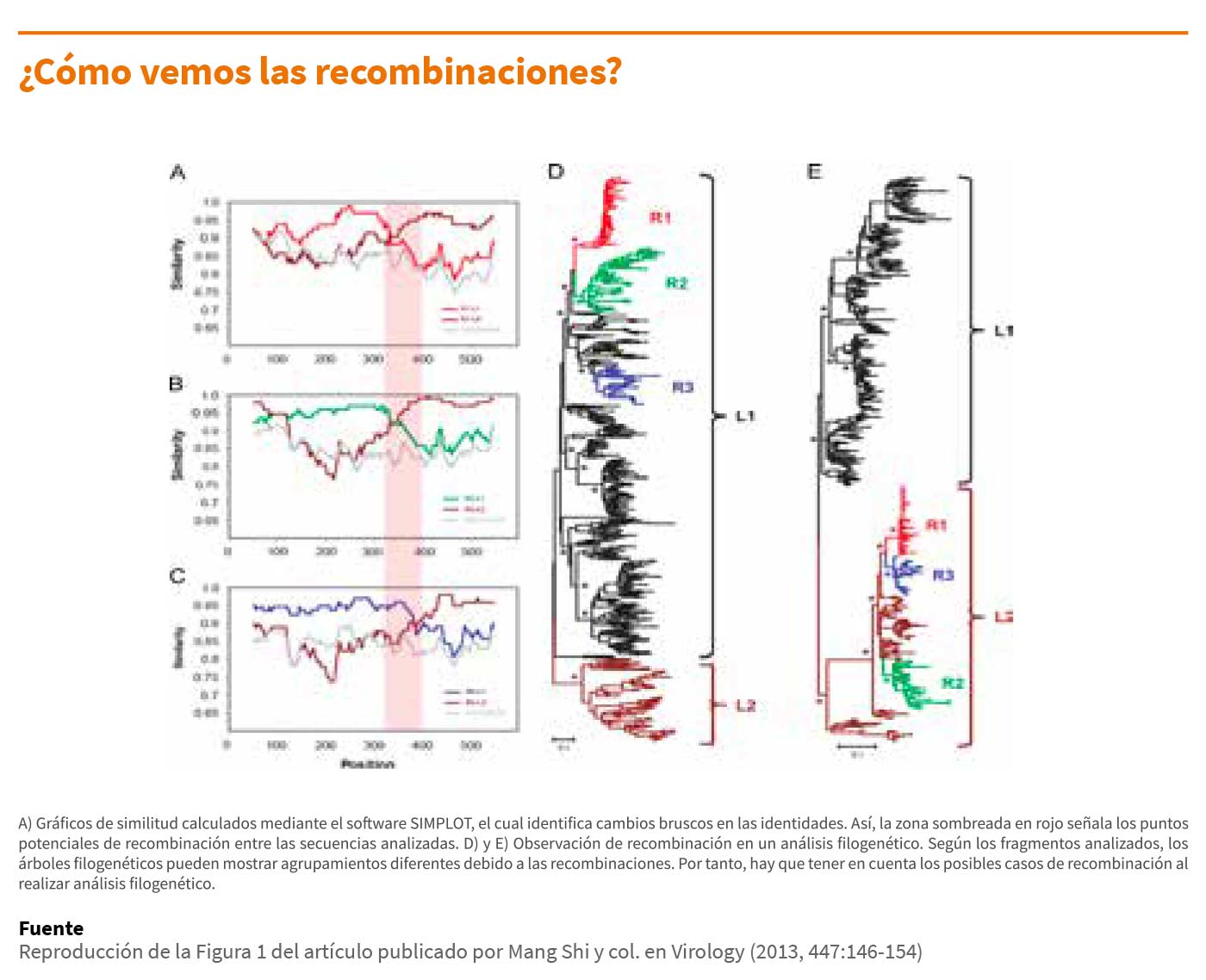
Los eventos de recombinación deben considerarse cuando se calculan los árboles filogenéticos, ya que pueden desdibujar las verdaderas relaciones filogenéticas entre cepas.
Secuenciación Sanger
Hasta hace poco, la secuenciación de Sanger ha sido la principal herramienta para estudiar la diversidad genética. Sin embargo, esta técnica tiene varias limitaciones. Una de las más importantes es que solo podemos obtener la secuencia de consenso.
Como se ha dicho en repetidas ocasiones, el virus del PRRS existe en el hospedador como cuasiespecie. Por lo tanto, los mutantes de baja frecuencia se obvian cuando se usa Sanger.
Además, las secuencias obtenidas mediante la técnica de Sanger son muy difíciles de interpretar si en la muestra hay presentes dos o más cepas.
Aunque representa una proporción muy limitada del genoma total (4%), la ORF5 se usa generalmente para comparar cepas. Esto es así porque esta ORF tiene una alta tasa de mutación y los cambios son fáciles de observar.
Obviamente, la secuenciación del genoma completo proporcionaría mucha más información:
- Agrupaciones filogenéticas y comparaciones entre cepas con mayor precisión
- Identificación de posibles epítopos, marcadores epidemiológicos o de virulencia
- Identificación de eventos de recombinación más allá de la ORF5.
Sin embargo, la secuenciación del genoma completo mediante Sanger es una tarea que requiere mucho tiempo, ya que precisa de la secuenciación de muchos segmentos de genoma superpuestos y consecutivos amplificados previamente por PCR.
En consecuencia, en comparación con ORF5 u ORF7, hay pocas secuencias de genoma completo disponibles.
Secuenciación masiva
Varias de las limitaciones que hemos discutido sobre secuenciación Sanger pueden superarse utilizando la secuenciación masiva o de próxima generación (NGS, por su acrónimo en inglés). El término se utiliza para describir una serie de diferentes tecnologías de secuenciación no basadas en Sanger.
Estas tecnologías tienen un enorme potencial y ciertamente son una revolución en el campo de la genómica y la biología molecular, ya que nos permiten secuenciar el ADN y el ARN mucho más rápido y de forma extremadamente más profunda que la secuenciación Sanger.
Ventajas de la NGS:
- Facilidad para obtener secuencias de genoma completo.
- Facilidad para determinar si hay más de una cepa en la muestra.
- Facilidad para identificar eventos de recombinación.
- Caracterización completa del espectro de mutantes (cuasiespecies). Es posible obtener de cientos a cientos de miles de secuencias a partir de una sola muestra.
Limitaciones de la NGS:
- La cantidad ingente de datos obtenidos de una sola muestra puede ser difícil de manejar.
- Se requiere elevada capacitación en bioinformática para evaluar las lecturas obtenidas y para minimizar la mala interpretación de los datos.
- Dificultad para discernir si una secuencia que muestra una mutación de baja frecuencia tiene un significado biológico.
- Hasta la fecha, el coste.
© Laboratorios Hipra, S.A. 2024. Reservados todos los derechos.
Ninguna parte de este sitio web o cualquiera de sus contenidos puede ser reproducida, copiada, modificada o adaptada, sin el consentimiento previo por escrito de HIPRA.
- Bautista EM, Meulenberg JJ, Choi CS, Molitor TW. Structural polypeptides of the American (VR-2332) strain of porcine reproductive and respiratory syndrome virus. Arch Virol. 1996:1357-65.
- Benfield DA, Nelson E, Collins JE, Harris L, Hennings JC, Shaw DP, Goyal SM, McCullough S, Morrison RB, Joo HS, Gorcyca D, Chladek D. Characterization of swine infertility and respiratory syndrome (SIRS) virus (isolate ATCC VR-2332). J Vet Diagn Invest. 1992. 4, 127-133.
- Benfield D, Nelson J, Rossow K, Nelson C, Steffen M, Rowland R. Diagnosis of persistent or prolonged porcine reproductive and respiratory syndrome virus infections Vet Res. 2000, 31:71.
- Brockmeier SL, Halbur PG, Thacker EL. Porcine respiratory disease complex. In KA Brogden, JM Guthmiller, eds, Polymicrobial Diseases. Washington, DC: ASM Press. 2002, 231-58.
- Calzada-Nova G, Schnitzlein W, Husmann R, Zuckermann FA. Characterization of the cytokine and maturation responses of pure populations of porcine plasmacytoid dendritic cells to porcine viruses and toll-like receptor agonists. Vet Immunol Immunopathol. 2010, 135:20-33.
- Chen WY, Schniztlein WM, Calzada-Nova G, Zuckermann FA. Genotype 2 strains of porcine reproductive and respiratory syndrome virus dysregulate alveolar macrophage cytokine production via the unfolded protein response. J Virol.2017, pii: JVI.01251-17.
- Christopher-Hennings J, Nelson EA, Hines RJ, Nelson JK, Swenson SL, Zimmerman JJ, Chase CL, Yaeger MJ, Benfield DA. Persistence of porcine reproductive and respiratory syndrome virus in serum and semen of adult boars. J Vet Diagn Invest. 1995, 7:456-64.
- Cortey M, Díaz I, Martín-Valls GE, Mateu E. Next-generation sequencing as a tool for the study of Porcine reproductive and respiratory syndrome virus (PRRSV) macro- and micro- molecular epidemiology. Vet Microbiol. 2017. doi: 10.1016/j.vetmic.2017.02.002.
- Darwich L, Díaz I, Mateu E. Certainties, doubts and hypotheses in porcine reproductive and respiratory syndrome virus immunobiology. Virus Res. 2010, 154:123-32.
- Díaz I, Darwich L, Pappaterra G, Pujols J, Mateu E. Different European-type vaccines against porcine reproductive and respiratory syndrome virus have different immunological properties and confer different protection to pigs. Virology. 2006, 351:249-59.
- Díaz I, Gimeno M, Darwich L, Navarro N, Kuzemtseva L, López S, Galindo I, Segalés J, Martín M, Pujols J, Mateu E. Characterization of homologous and heterologous adaptive immune responses in porcine reproductive and respiratory syndrome virus infection. Vet Res. 2012, 19:43:30.
- García-Nicolás O, Quereda JJ, Gómez-Laguna J, Salguero FJ, Carrasco L, Ramis G, Pallarés FJ. Cytokines transcript levels in lung and lymphoid organs during genotype 1 Porcine Reproductive and Respiratory Syndrome Virus (PRRSV) infection. Vet Immunol Immunopathol. 2014, 160:26-40.
- Gimeno M, Darwich L, Diaz I, de la Torre E, Pujols J, Martín M, Inumaru S, Cano E, Domingo M, Montoya M, Mateu E. Cytokine profiles and phenotype regulation of antigen presenting cells by genotype-I porcine reproductive and respiratory syndrome virus isolates. Vet Res. 2011, 42:9.
- Gómez-Laguna J, Salguero FJ, Pallarés FJ, Carrasco L. Immunopathogenesis of porcine reproductive and respiratory syndrome in the respiratory tract of pigs. Vet J. 2013, 195:148-55.
- Haynes JS, Halbur PG, Sirinarumitr T, Paul PS, Meng XJ, Huffman EL. Temporal and morphologic characterization of the distribution of porcine reproductive and respiratory syndrome virus (PRRSV) by in situ hybridization in pigs infected with isolates of PRRSV that differ in virulence. Vet Pathol. 1997, 34:39-43.
- Hill H. Overview and history of Mystery Swine Disease (swine infertility/respiratory syndrome). Proceedings of the Mystery Swine Disease Committee Meeting, Livestock Conversation Institute, Denver, CO. 1990, 29–31.
- Holtkamp DJ, Polson DD, Torremorrell M. Terminology for classifying swine herds by porcine reproductive and respiratory status. J Swine Health and Prod. 2011, 19:44-56.
- Horter DC, Pogranichniy RM, Chang CC, Evans RB, Yoon KJ, Zimmerman JJ. Characterization of the carrier state in porcine reproductive and respiratory syndrome virus infection. Vet Microbiol. 2002, 86:213-28.
- Loula T. Mystery pig disease. Agri-practice. 1991, 12:23–34.
- Lunney JK, Benfield DA, Rowland RR. Porcine reproductive and respiratory syndrome virus: an update on an emerging and re-emerging viral disease of swine. Virus Res. 2010, 154:1-6.
- Mateu E, Tello M, Coll A, Casal J, Martín M. Comparison of three ELISAs for the diagnosis of porcine reproductive and respiratory syndrome. Vet Rec. 2006, 159:717-8.
- Mateu E, Diaz I. The challenge of PRRS immunology. Vet J. 2008, 177:345-51.
- Meier WA, Galeota J, Osorio FA, Husmann RJ, Schnitzlein WM, Zuckermann FA. Gradual development of the interferon-gamma response of swine to porcine reproductive and respiratory syndrome virus infection or vaccination. Virology. 2003, 309:18-31.
- Nelson EA, Christopher-Hennings J, Benfield DA. Serum immune responses to the proteins of porcine reproductive and respiratory syndrome (PRRS) virus. J Vet Diagn Invest. 1994, 6:410-5.
- Appendices IV and V of the REPORT OF THE OIE AD HOC GROUP ON PORCINE REPRODUCTIVE RESPIRATORY SYNDROME Paris, 9 – 11 June 2008 http://www.oie.int/fileadmin/Home/eng/Our_scientific_expertise/docs/pdf/PRRS_guide_web_bulletin.pdf.
- Rodríguez-Gómez IM, Gómez-Laguna J, Carrasco L. Impact of PRRSV on activation and viability of antigen presenting cells. World J Virol. 2013, 2:146-51.
- Rovira A, Cano JP, Muñoz-Zanzi C. Feasibility of pooled-sample testing for the detection of porcine reproductive and respiratory syndrome virus antibodies on serum samples by ELISA. Vet Microbiol. 2008, 130:60-8.
- Rovira A, Clement T, Christopher-Hennings J, Thompson B, Engle M, Reicks D, Muñoz-Zanzi C. Evaluation of the sensitivity of reverse-transcription polymerase chain reaction to detect porcine reproductive and respiratory syndrome virus on individual and pooled samples from boars. J Vet Diagn Invest. 2007, 19:502-9.
- Rovira A, Reicks D, Muñoz-Zanzi C. Evaluation of surveillance protocols for detecting porcine reproductive and respiratory syndrome virus infection in boar studs by simulation modeling. J Vet Diagn Invest. 2007, 19:492-501.
- Salguero FJ, Frossard JP, Rebel JM, Stadejek T, Morgan SB, Graham SP, Steinbach F. Host-pathogen interactions during porcine reproductive and respiratory syndrome virus 1 infection of piglets. Virus Res. 2015, 202:135-43.
- Segalés J, Domingo M, Balasch M, Solano GI, Pijoan C. Ultrastructural study of porcine alveolar macrophages infected in vitro with porcine reproductive and respiratory syndrome (PRRS) virus, with and without Haemophilus parasuis. J Comp Pathol. 1998, 118:231-43.
- Sur JH, Cooper VL, Galeota JA, Hesse RA, Doster AR, Osorio FA. In vivo detection of porcine reproductive and respiratory syndrome virus RNA by in situ hybridization at different times postinfection. J Clin Microbiol. 1996, 34:2280-6.
- Terpstra C, Wensvoorst G, Pol JMA. Experimental reproduction of porcine epidemic abortion and respiratory syndrome (mystery swine disease) by infection with Lelystad virus: Koch’s postulates fulfilled. Vet Q. 1991, 13:131–36.
- Tian K, Yu X, Zhao T, Feng Y, Cao Z, Wang C, Hu Y, Chen X, Hu D, Tian X, Liu D, Zhang S, Deng X, Ding Y, Yang L, Zhang Y, Xiao H, Qiao M, Wang B, Hou L, Wang X, Yang X, Kang L, Sun M, Jin P, Wang S, Kitamura Y, Yan J, Gao GF. Emergence of fatal PRRSV variants: unparalleled outbreaks of atypical PRRS in China and molecular dissection of the unique hallmark. PLoS One. 200, 2:e526.
- Tong GZ, Zhou YJ, Hao XF, Tian ZJ, An TQ, Qiu HJ. Highly pathogenic porcine reproductive and respiratory syndrome, China. Emerg Infect Dis. 2007, 13:1434-6.
Van Alstine WG, Popielarczyk M, Albregts SR. Effect of formalin fixation on the immunohistochemical detection of PRRS virus antigen in experimentally and naturally infected pigs. J Vet Diagn Invest. 2002, 14:504-7. - Vézina SA, Loemba H, Fournier M, Dea S, Archambault D. Antibody production and blastogenic response in pigs experimentally infected with porcine reproductive and respiratory syndrome virus. Can J Vet Res. 1996, 60:94-9.
- Wensvoort G, Terpstra C, Pol JMA, Lask EA, Bloemraad M, de Kluyver EP, Kragten C, van Butten L, den Besten A, Wagenaar F, Broekhuijsen JM, Moonen PJM, Zetstra T, de Boer EA, Tibben AhJ, de Jong MF, van’r Veld P, Groenland GJR, van Gennep JA, Voets MTh, Verheijden JHM, Braamkamp J. Mystery swine disease in the Netherlands: the isolation of Lelystad virus. Vet Q. 1991, 13:121–30.
- Wills RW, Doster AR, Galeota JA, Sur JH, Osorio FA. Duration of infection and proportion of pigs persistently infected with porcine reproductive and respiratory syndrome virus. J Clin Microbiol. 2003, 41:58-62.
- Zimmerman JJ, Benfield DA, Dee SA, Murtaugh MP, Stadejek T, Stevenson GW, Torremorell M. Porcine reproductive and respiratory syndrome virus (porcine arterivirus). In: 10th ed. Diseases of swine, Ed. Wiley-Blackwell. 2012, 31:463-86.


 Todos los derechos reservados. © HIPRA
Todos los derechos reservados. © HIPRA
