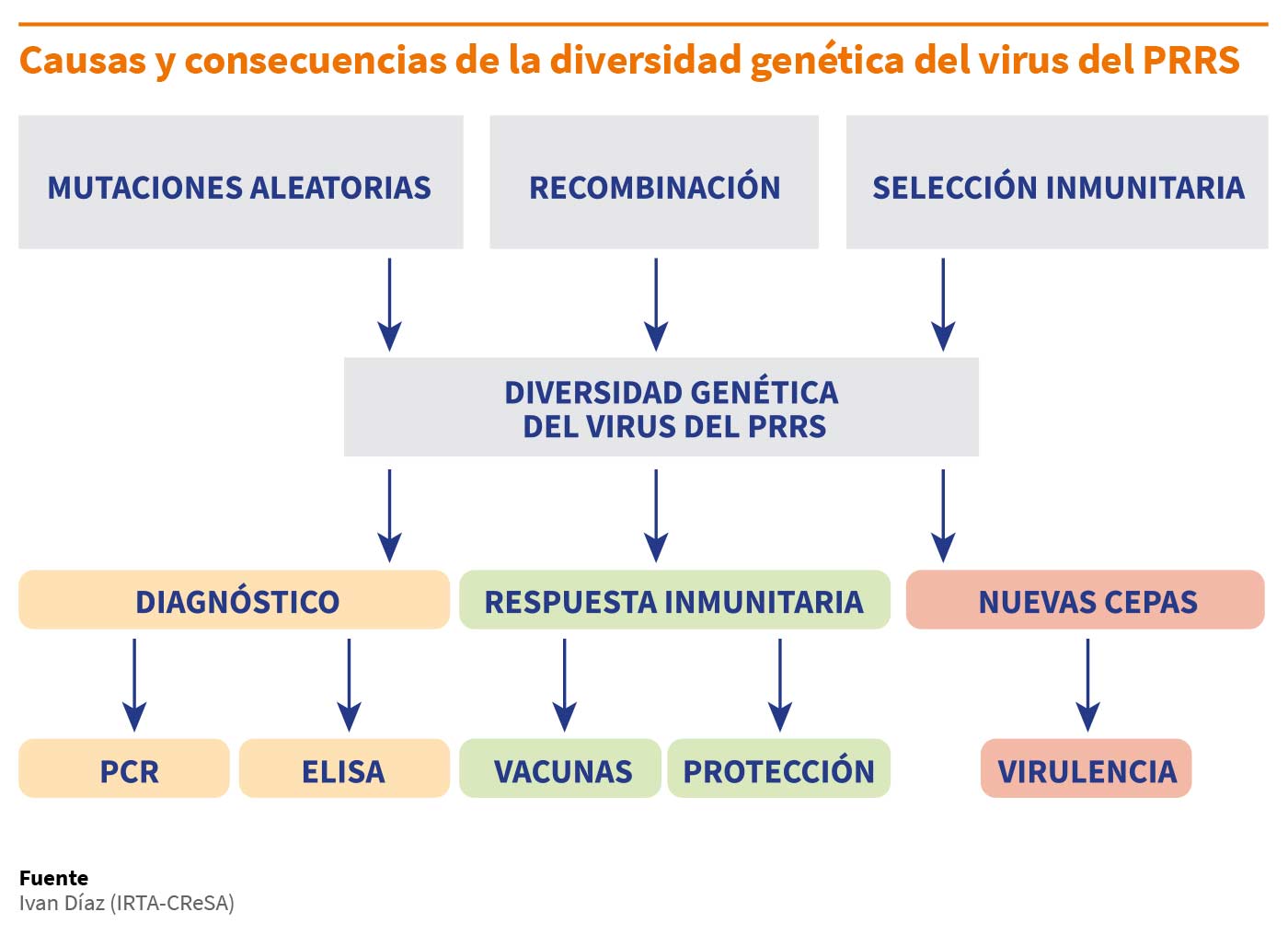
La diversidad genética del virus del PRRS es el resultado de los fenómenos de mutación y selección, pero también de recombinación:
- Mutaciones aleatorias: Asumimos que la tasa de mutación del virus del PRRS es una de las más altas, siendo hasta 40 veces superior a la tasa de mutación de otros virus muy conocidos, como el virus de la Influenza Aviar o el virus de la Inmunodeficiencia Humana.
Como ocurre con otros virus ARN, su ARN polimerasa no tiene la capacidad de corregir los errores comunes e inherentes que ocurren durante la transcripción. Dado que estos errores aparecen cada 100-1.000 nucleótidos, cada nuevo virus que se forma puede ser diferente al anterior. Como consecuencia, se producen nuevas variantes virales que tienen secuencias nucleotídicas diferentes a las del progenitor. Así, en un cerdo infectado, el virus del PRRS existe como una nube, agrupación o distribución de variantes víricas diversas, las cuales están relacionadas entre sí por tener mutaciones similares. Las variantes contribuyen colectivamente a las características de la población y están sujetas a la variación, competición y selección. Esta forma de distribución se conoce como cuasiespecie. Se ha especulado que la inmunomodulación ejercida por parte del virus y la evolución en cuasiespecies juegan un papel muy importante durante la infección del virus del PRRS y su curso crónico o de persistencia. - Recombinación: (intercambio de material genético entre dos o más cepas). En algunos casos, las diferencias entre cepas son tan evidentes que aceptar que proceden unicamente de mutaciones es imposible.
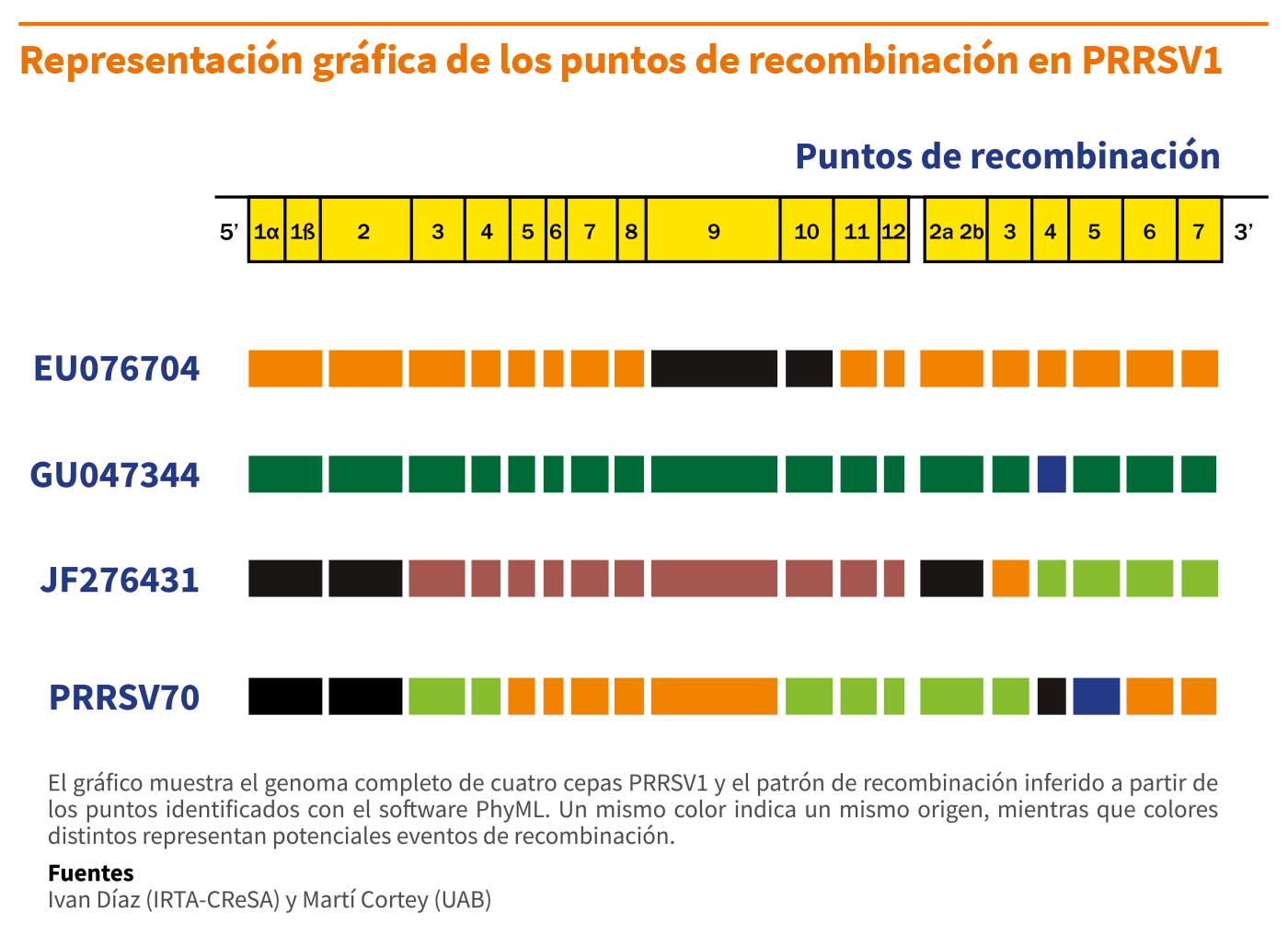
Además, las mutaciones por sí solas tampoco pueden explicar la presencia de deleciones e inserciones en numerosas localizaciones a lo largo del genoma. De hecho, cuando se examina el genoma completo en ambas especies, se detectan frecuentemente eventos de recombinación. Por tanto, podemos afirmar que la recombinación es un fenómeno muy importante que también nos ayuda a entender la elevada diversidad genética del virus.

 Todos los derechos reservados. © HIPRA
Todos los derechos reservados. © HIPRA
